R语言怎么做相关性分析
- 教育
- 40秒前
- 586

R语言怎么做相关性分析
1、相关性分析相关性分析是指对两个或多个具备相关性的变量元素进行分析,从而衡量两个变量因素的相关密切程度。相关性分析旨在研究两个或两个以上随机变量之间相互依存关系的方向和密切程度。一般来讲研究对象(样品或处理组)之间使用距离分析,而元素(物种或环境因子)之间进行相关性分析。两个变量之间的相关性可以用简单相关系数(例如皮尔森相关系数等)进行表示,相关系数越接近1,两个元素相关性越大,相关系数越接近0,两个元素越独立。
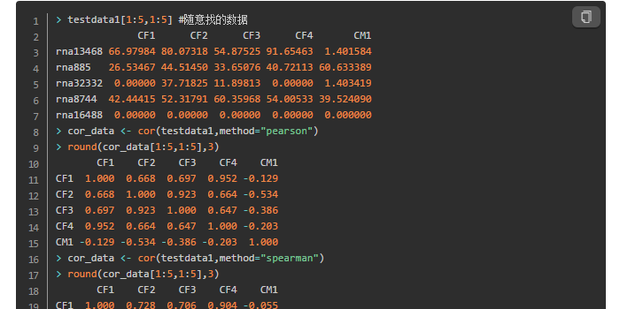
2、相关性指标(1)Pearson相关系数(皮尔逊积差相关系数)Pearson相关系数是用于表示相关性大小的最常用指标,数值介于-1~1之间,越接近0相关性越低,越接近-1或1相关性越高。正负号表明相关方向,正号为正相关、负号为负相关。适用于两个正态分布的连续变量。(2)Spearman等级相关系数(斯皮尔曼秩相关系数)利用两变量的秩次大小来进行分析,属于非参数统计方法。适用于不满足Pearson相关系数正态分布要求的连续变量。也可以用于有序分类变量的之间的相关性测量。(3)Kendall's Tau相关系数Kendall's Tau相关系数是一种非参数检验,适用于两个有序分类变量。(4)其它此外衡量两个变量之间关系的方法还有:卡方检验、Fisher精确检验等。cor函数Pearson、Spearman、Kendall相关系数都可以通过cor函数实现,cov协方差函数参数同
3、cor函数。(1)用法x:矩阵或数据框。use:指定缺失数据的处理方式。可选项:all.obs(假设不存在缺失数据)、everything(数据存在缺失值时,相关系数计算结果会显示missing)、complete.obs(行删除)、pairwise.complete.obs(成对删除)。method:指定相关系数的类型。可选类型为pearson、spearman、kendall。(2)R Scriptggcorrplotggcorrplot包内只有2个函数,一个cor_pmat()用于计算p值,一个ggcorrplot()用于绘图。ggcorrplot相当于精简版的corrplot包,只有主题更加丰富多样。
4、样品间相似性(similarity)和距离(distance)(1)表示距离的方法欧式距离(Euclidean Distance)√(a2+b2+c^2)dist(t(x),p=2)曼哈顿距离(Manhattan Distance)|a1-a2|+|b1-b2|+|c1-c2|dist(t(x),"manhattan")切比雪夫距离(Chebyshev Distance)max(|a1-a2|,|b1-b2|,|c1-c2|)dist(t(x),"maximum")闵可夫斯基距离(Minkowski Distance)dist(t(x),"minkowski")标准化欧氏距离(Standardized Euclidean distance)先将数据各维分量标准化到均值方差相等,即(x-μ)/δ,标准化后的值=(标准化前的值-分量的均值)/分量的标准差。x1 = scale(t(x), center=T,scale=T)dist(x1)马氏距离(Mahalanobis Distance)兰式距离dist(t(x), method = "canberra")夹角余弦(Cosine)汉明距离(Hamming distance)两个等长字符串s1与s2之间的汉明距离定义为将其中一个变为另外一个所需要作的最小替换次数。
5、x <- c(1, 0, 0)y <- c(1, 0, 1)hamming.distance(x, y) #1杰卡德相似系数(Jaccard similarity coefficient)dist(t(x), method = "Jaccard")相关系数(Correlation coefficient)与相关距离(Correlation distance)1-cor(x)信息熵(Information Entropy)
6、信息熵是衡量分布的混乱程度或分散程度的一种度量。分布越分散(分布越平均),信息熵就越大。分布越有序(分布越集中),信息熵就越小。kl散度(2)dist用法This function computes and returns the distance matrix computed by using the specified distance measure to compute the distances between the rows of a data matrix.这个函数用特定的方法计算矩阵的行之间的距离,并返回距离矩阵。dist(x, method = "euclidean", diag = FALSE, upper = FALSE, p = 2) method:可以是"euclidean", "maximum", "manhattan", "canberra", "binary", "minkowski"diag:是否显示对角线的值upper:是否显示上三角的值p:The power of the Minkowski distance(3)scale用法scale(x, center = TRUE, scale = TRUE)
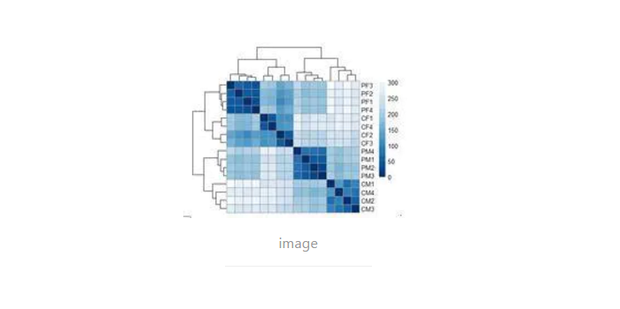
7、scale是对矩阵的每一列进行标准化,如果要对行标准化需要先转置。如heatmapdata <- t(scale(t(heatmapdata)))(4)R ScriptsampleDist <- dist(t(testdata1)) sampleDistMatrix <- as.matrix(sampleDist) colnames(sampleDistMatrix) <- NULL colors <- colorRampPalette(rev(brewer.pal(9,"Blues")))(255) pheatmap(sampleDistMatrix, clustering_distance_rows=sampleDist, clustering_distance_cols=sampleDist, color = colors)